코드 링크:
MedImagingDL/ch05_06_3d_brain_tumor_segmentation_infe.ipynb at 55edfd5ed184027a708c9aa1fdf732680390de3f · ahxlzjt/MedImagingDL
#MedicalDL #CV #AI . Contribute to ahxlzjt/MedImagingDL development by creating an account on GitHub.
github.com
0. 라이브러리 설치 및 임포트
- !pip install monai, !pip install natsort로 필요 라이브러리 설치
- MONAI, PyTorch, NumPy, Matplotlib 등을 불러옴
1. Config 정의 및 Dataset 준비
1.1 cfg 클래스
- base_path, train_path, test_path 등 프로젝트 경로 지정
- seed, max_epochs, in_channels=4, out_channels=3 설정
1.2 train_data_dicts, test_data_dicts 생성
- Brats 2020 데이터 폴더를 glob하여,
- 5개 파일(*_flair.nii, *_t1.nii, *_t1ce.nii, *_t2.nii, *_seg.nii)이 있으면 train으로
- 4개 파일(*_flair.nii, *_t1.nii, *_t1ce.nii, *_t2.nii)이 있으면 test로
- 각각 {'image': [...], 'label': [...]} 형태 딕셔너리로 저장
2. Custom Transform: ConvertToMultiChannelBasedOnBratsClassesd
- Brats의 label은 {0,1,2,3} (배경=0, ED=1, ET=2, NET/NCR=3)
- 이 클래스를 TC, WT, ET로 나누어 3채널로 변환
- TC(Tumor Core): label 2 or 3
- WT(Whole Tumor): label 1 or 2 or 3
- ET(Enhancing Tumor): label 2만
3. Data Transforms (Train/Val)
3.1 Train Transform
- LoadImaged, EnsureChannelFirstd: 4채널 이미지를 로드
- ConvertToMultiChannelBasedOnBratsClassesd: 위에서 정의한 3채널 라벨로 매핑
- Orientationd(axcodes="RAS"), Spacingd(pixdim=(1,1,1)): 방향·스페이싱 정규화
- RandSpatialCropd(roi_size=[224,224,144]): 고정 크기 크롭
- NormalizeIntensityd(채널단위로 Z-score 등)
- RandScaleIntensityd(factors=0.1): 강도 스케일링
3.2 Val Transform
- 위와 거의 동일하나, Rand형 변환(augment)은 제외
4. Dataset Split & Loader
- train_dict, valid_dict = train_test_split(train_data_dicts, test_size=0.2, random_state=2023)
- train_dataset = Dataset(train_dict, transform=train_transform)
- train_loader = DataLoader(..., batch_size=1, shuffle=True, ...)
- valid_dataset = Dataset(valid_dict, transform=val_transform)
- val_loader = DataLoader(..., batch_size=1, shuffle=False, ...)
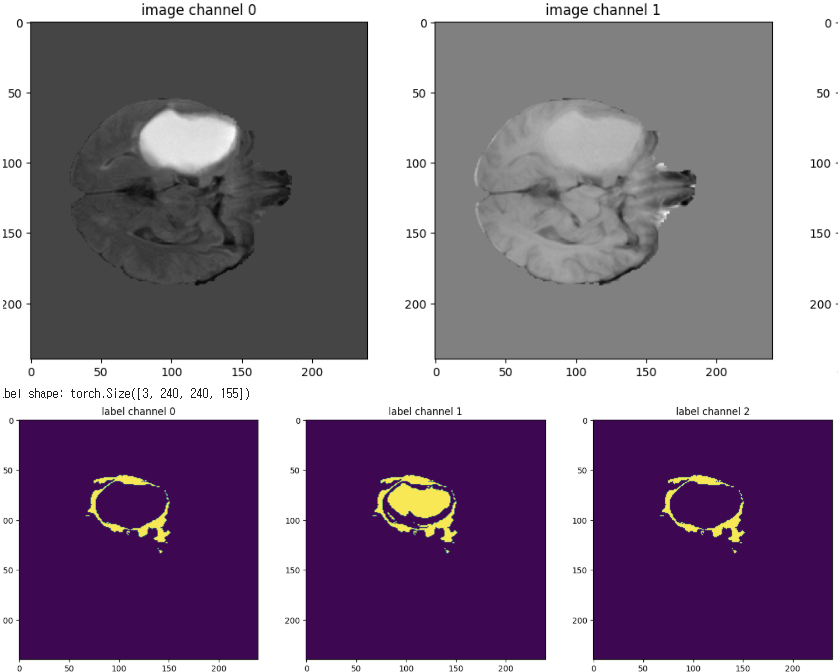
5. Visualization
- val_data_example = valid_dataset[2]
- 이미지 shape, label shape 출력
- plt.imshow(val_data_example["image"][i, :, :, 60]) 등으로 채널별 슬라이스 시각화
- 라벨도 3채널로 각각 확인

6. Seed 고정 & Model 초기화
- seed_everything(cfg.seed)
- 랜덤 시드 고정 (PyTorch, NumPy 등)
- SegResNet 모델 생성
- model = SegResNet(...) with in_channels=4, out_channels=3
- Loss/Optimizer 설정
- loss_function = DiceLoss(...)
- optimizer = torch.optim.Adam(...)
- lr_scheduler = CosineAnnealingLR(...)
- DiceMetric 준비
- dice_metric = DiceMetric(...)
- dice_metric_batch = DiceMetric(...) (배치별 평균 계산)
7. Inference 함수 정의
- sliding_window_inference 사용
- val_AMP=True 시 torch.cuda.amp.autocast로 연산 가속
8. 학습 루프
- 모델 훈련(model.train())
- DataLoader 반복
- with torch.cuda.amp.autocast(): outputs = model(inputs)
- loss_function(outputs, labels)
- AMP 스케일링(scaler.scale(loss).backward(), scaler.step(optimizer))
- 일정 스텝마다 train_loss 출력
- lr_scheduler.step()로 학습률 업데이트
- 검증(model.eval())
- inference + post-processing(Activations+AsDiscrete)
- dice_metric에 추가
- TC, WT, ET 각각 dice 출력
- best_metric 갱신 시 모델 weight 저장(best_metric_model.pth)

9. 학습 결과 시각화

- plt.plot(epoch_loss_values)로 Epoch Average Loss
- plt.plot(metric_values)로 Val Mean Dice 표시
- 채널별(TC, WT, ET) 그래프도 추가로 그려서 성능 추이 확인
10. Inference on a sample

- model.load_state_dict(torch.load("best_metric_model.pth"))
- val_input = valid_dataset[2]["image"].unsqueeze(0).cuda()
- val_output = inference(val_input)
- val_output = post_trans(val_output[0])
- 슬라이스 70번 등 임의로 골라 원본 이미지, 라벨, 모델 출력 각각 시각화
11. 정리
- Dataset: Brats multi-modal(4채널) + 라벨(1채널) → 변환을 통해 3채널 분류(TC, WT, ET)
- SegResNet 모델 기반 DiceLoss 학습
- sliding_window_inference로 3D 세그멘테이션 수행
- Batch=1로 GPU 메모리 절약, AMP로 연산 가속
- DiceMetric으로 매 epoch 검증 & 최적모델 저장
- 최종적으로 Validation 예시를 그림으로 확인