논문 링크: https://arxiv.org/pdf/2409.11169v2
OUTTA 논문 리뷰 링크: [2025-1] 유경석 - MAISI: Medical AI for Synthetic Imaging
[2025-1] 유경석 - MAISI: Medical AI for Synthetic Imaging
https://arxiv.org/pdf/2409.11169v2 https://build.nvidia.com/nvidia/maisi maisi Model by NVIDIA | NVIDIA NIMMAISI is a pre-trained volumetric (3D) CT Latent Diffusion Generative Model.build.nvidia.com AbstractMAISI (Medical AI for Synthetic Imaging) : 3D
blog.outta.ai
1. Introduction
의료 영상 ML 모델 개발의 한계점
- 데이터 희소성: 희귀 질환 데이터 부족 → 모델 학습 한계
- Human annotation 비용: 정확한 진단을 위한 전문 지식 필요 → 비용 상승
- Privacy 문제: 환자 정보 보호 필요 → 윤리적 문제 발생
- Synthetic data 생성 필요성: 의료 이미지의 인공적 생성 → Data augmentation, 환자 데이터 의존성 감소, cost-effective annotation 가능
기존 연구 한계점
- 고해상도 3D volume 생성의 어려움: 3D framework의 높은 메모리 사용량 → Memory bottleneck 해결 필요
- 고정된 output volume dimensions, voxel spacing: 다양한 작업에 대한 유연성 부족
- 특정 데이터/장기 기반 모델: 일반화 불가 → 다양한 조건에서 retraining 필요
제안하는 모델
- Volume Compression Network: 대량의 3D 의료 데이터를 latent space로 압축 → 메모리 절약
- Latent Diffusion Model: Flexible dimension 및 Condition(Body region, voxel spacing) 설정 → 낮은 메모리 소비로 복잡한 구조 생성
- ControlNet: Output 제어 → 다양한 작업 적용 가능, retraining 필요성 감소

2. Related Work
기존 의료 영상 생성 방법
- Example-based approach, geometry-regularized dictionary learning → 제한된 생성 능력
Deep learning 기반 생성 모델
- GAN
- MRI/CT 이미지 합성, cross-modality image translation, image reconstruction, super-resolution 활용
- 한계: 3D complexity 고려 부족 → 2D medical image 및 작은 volumetric patch synthesis에 한정
- Diffusion Model (DM)
- 고품질 영상 합성, 안정적 학습과정, conditioning 유연성 제공
- GenerateCT: 3D CT volume 합성 → slice 단위 생성 → slice 간 3D 구조 불일치 문제
- DiffTumor: Organ tumor segmentation 모델의 robustness, generalizability 향상
3. Methodology
3.1 Volume Compression Network (VAE-GAN)
- VAE 적용: Combined objectives로 학습
- Perceptual loss $L_{lips}$
- Adversarial loss $L_{adv}$
- L1 reconstruction loss $L_{recon}$
- KL regularization $L_{reg}$: High-variance latent space 방지


- Tensor Splitting Parallelism (TSP): Memory bottleneck 해결
- 기존 문제: Super-resolution model → GPU memory limitation
- Sliding window inference: 3D patch stitching 시 boundary artifacts 문제 발생
- TSP 해결책: Feature map을 segment로 분할 후 지정된 device에 병렬 할당 → 메모리 사용량 최적화

3.2 Latent Diffusion Model
- 압축된 latent space에서 동작
- Conditional inputs: Body region, voxel spacing → Flexible dimension 설정 가능
- Denoising 과정 학습: Markov chain 기반 → Time-conditional U-Net 사용
3.3 Additional Conditioning Mechanisms
- 생성된 output 제어 및 flexibility 향상
- Locked copy: Original model knowledge 보존
- Trainable copy: 특정 조건에 맞춰 학습 가능
- Zero convolution layer: Compact encoder network 활용 → Task-specific condition $c_f$ 적용
4. Experiments
4.1 Datasets and Implementation Details
- 3개 모델 (VAE, DM, ControlNet) 학습 → 정상 범위 내 주요 장기 생성 여부 평가
4.2 Evaluation of MAISI VAE
- Out-of-distribution dataset 실험: MAISI VAE vs. Dedicated VAE 비교
- 결과: 추가 GPU 자원 없이 유사한 성능 달성 → 모델 효율성과 실용성 입증
4.3 Evaluation of MAISI Diffusion Model
- Synthesis quality 평가
- 실제 dataset과 비교하여 FID score 측정
- DDPM, LDM, HA-GAN 대비 실제 dataset과 유사한 CT 이미지 생성

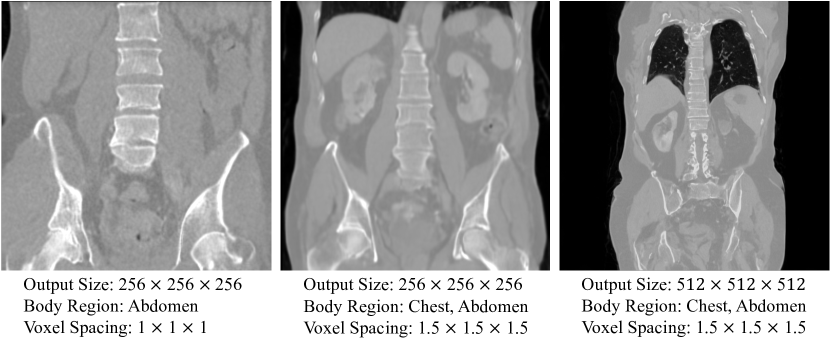
- Response to primary conditions
- Body region, voxel spacing condition 반영 → 높은 flexibility, control 제공
4.4 Data Augmentation in Downstream Tasks

- Segmentation model의 DCS (Dice Similarity Coefficient) 비교
- Real Only dataset vs. Real + Synthetic dataset
- MAISI CT Generation: 일반적인 CT 이미지 생성
- MAISI Inpainting: 건강한 환자 데이터에 tumor 합성
- 결과: MAISI dataset augmentation을 통한 segmentation 성능 향상
- Out-of-distribution dataset에서도 높은 성능 유지
5. Discussion and Limitation
- 장점
- 고품질 CT 이미지 생성 가능
- 다양한 임상 시나리오에서 적용 가능
- 한계점
- 인구 통계적 변화 표현 부족
- 계산 리소스 소모 여전히 큼
6. Conclusion
- VAE + LDM 기반의 Foundation Model과 ControlNet 조합
- 고해상도 3D CT Volume 생성 가능
- 해부학적으로 정확한 이미지 생성
- Flexible volume dimensions 및 voxel spacing 제공
- 의료 데이터 증강을 통해 Downstream task 성능 향상
더보기
MAISI 논문 관련 Q&A
Q1. FID, MSD Task 06, LIDC-IDRI, TCIA란? 의료 영상에서는 어떻게 측정하나?
- FID (Fréchet Inception Distance)
- 실제 데이터와 생성된 데이터 간의 feature distribution 차이를 측정하는 지표
- 의료 영상에서는 Inception Network 대신 Medical Feature Extractor 사용 가능
- MSD Task 06
- Medical Segmentation Decathlon 챌린지의 Task 06 (Lung Tumor Segmentation)
- 폐 종양 분할을 위한 데이터셋
- LIDC-IDRI
- Lung Image Database Consortium - Image Database Resource Initiative
- CT 기반 폐 결절(결절성 종양) 데이터셋
- TCIA (The Cancer Imaging Archive)
- 다양한 암 관련 의료 영상 데이터를 제공하는 오픈 데이터베이스
Q2. 3D 의료 이미지(예: a.nii, b.nii, c.nii)의 FID Score 계산 코드
import torch
import nibabel as nib
import numpy as np
from torchmetrics.image.fid import FrechetInceptionDistance
from monai.transforms import Compose, EnsureChannelFirst, Resize
# 데이터 전처리
def load_nifti(file_path, target_shape=(128, 128, 128)):
img = nib.load(file_path).get_fdata()
img = np.clip(img, np.percentile(img, 1), np.percentile(img, 99)) # intensity normalization
img = img / np.max(img) # Normalize
img = torch.tensor(img, dtype=torch.float32)
img = img.unsqueeze(0) # Add channel dimension
transform = Compose([EnsureChannelFirst(), Resize(target_shape)])
return transform(img)
# FID 계산
def calculate_fid(real_paths, generated_paths):
fid = FrechetInceptionDistance(feature=64) # 사용할 feature layer 설정
real_images = torch.stack([load_nifti(p) for p in real_paths])
generated_images = torch.stack([load_nifti(p) for p in generated_paths])
fid.update(real_images, real=True)
fid.update(generated_images, real=False)
return fid.compute()
# 사용 예시
real_images = ["real_a.nii", "real_b.nii"]
generated_images = ["gen_a.nii", "gen_b.nii"]
fid_score = calculate_fid(real_images, generated_images)
print(f"FID Score: {fid_score}")Q3. 각 plane별 FID 비교 이유? (DDPM, LDM, HA-GAN, MAISI)
- 3D 의료 영상은 다양한 방향에서 다르게 보일 수 있음
- XY, XZ, YZ plane별로 FID 비교
→ 특정 방향에서의 생성 품질 차이를 분석하기 위함
→ 일관된 구조를 유지하는지 평가
Q4. LDM vs. MAISI 차이점
- LDM (Latent Diffusion Model)
- Pixel space가 아닌 latent space에서 diffusion 적용 → 메모리 효율적
- 고정된 output shape 사용
- MAISI (Medical AI for Synthetic Imaging)
- LDM 기반이지만 의료 영상 특화된 설계
- Flexible dimension 제공 (Body region, voxel spacing 반영)
- ControlNet 추가 적용 → 다양한 조건에서 retraining 없이 사용 가능
Q5. MAISI의 이미지 shape 변화 과정?
- 예시: (512, 512, 768) full CT image
→ Patch로 자르면 (1, 64, 64, 64)
→ Batch 처리 시 (4 × 64 × 64 × 64)
→ AutoEncoder 통과 후 (8, 8, 40, 16) - 다양한 output size 조합 가능 (GitHub 참고)
Q6. DDPM, HA-GAN은 AutoEncoder 없이 pixel space에서 진행? GPU 연산량 차이?
- DDPM, HA-GAN
- AutoEncoder 없이 pixel space에서 진행
- 메모리 사용량 ↑ → 3D medical image에서는 연산량 매우 큼
- MAISI vs. DDPM, HA-GAN GPU 요구량
- MAISI는 latent space에서 diffusion 진행 → 연산량 감소
- DDPM, HA-GAN: 수십 GB GPU 필요
- MAISI: 메모리 절약 가능 (VAE-GAN + TSP 사용)
Q7. MAISI가 일반적인 LDM보다 나은 점 3가지
- Flexible volume dimensions 제공 → 다양한 의료 작업에 적용 가능
- ControlNet 적용 → 재학습 없이 다양한 조건 반영 가능
- 메모리 최적화 (VAE-GAN + TSP 적용) → 3D CT 데이터 효율적 처리 가능
Q8. MONAI에 DDPM, LDM, HA-GAN 코드가 존재하나?
- DDPM: 🔗 MONAI 3D DDPM
- LDM: 🔗 MONAI 3D LDM
- HA-GAN: 🔗 HA-GAN GitHub
Q9. MAISI VAE vs. Dedicated VAE vs. VQVAE 차이
- MAISI VAE
- 일반적인 VAE 구조 + adversarial loss 적용
- Medical 데이터에 최적화됨
- Dedicated VAE
- 특정 dataset 전용 VAE
- 일반화 능력 낮음
- VQVAE (Vector Quantized VAE)
- Discrete latent space 사용
- 코드북 (codebook) 기반 압축
Q10. MAISI 논문의 DDPM은 3D 의료 영상 전용인가?
- Yes
- 3D 의료 영상 생성을 위해 설계된 DDPM
Q11. MAISI AutoEncoder 학습 방식? (MRI + Condition 포함?)
- (B) MRI + 3D Seg로 학습
- MRI와 3D segmentation을 함께 사용
- Conditioning을 위해 segmentation 정보도 활용
Q12. Segmentation image를 condition으로 넣을 때 AutoEncoder를 사용해 차원 축소하나?
- Yes, AutoEncoder 사용
- (A) MRI로만 학습한 AE → 3D segmentation 데이터 압축 시 정보 손실 가능
- (B) MRI + 3D Seg로 학습한 AE → 의미 있는 feature 보존 가능
- (C) 3D Seg만 학습한 AE → MRI 재구성 능력 부족
결론: (B) MRI + 3D Seg 학습 방식이 최적