논문 링크: 1809.05231
OUTTA 논문 리뷰 링크: [2024-2] 백승우 - VoxelMorph: A Learning Framework for Deformable Medical Image Registration
[2024-2] 백승우 - VoxelMorph: A Learning Framework for Deformable Medical Image Registration
VoxelMorph: A Learning Framework for Deformable Medical Image RegistrationWe present VoxelMorph, a fast learning-based framework for deformable, pairwise medical image registration. Traditional registration methods optimize an objective function for each p
blog.outta.ai
0. Abstract
VoxelMorph은 변형 가능한 pair별 의료 image registration을 위한 빠른 learning-based framework임.
Image Registration (영상 정합)
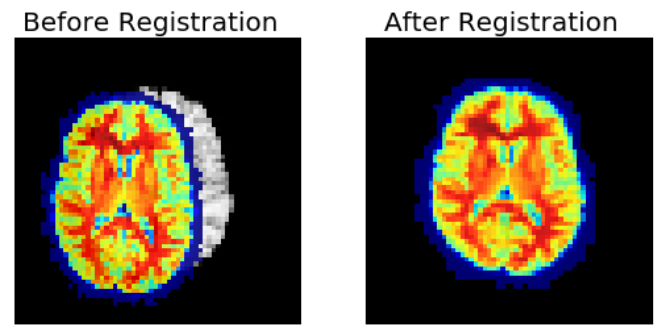
서로 다른 좌표계를 가진 두 영상을 동일한 좌표계로 정렬하는 과정이며, 이를 위해 맵핑되는 기하학적 변환을 찾는 과정임.
Voxel-based Registration (복셀 기반 정합)
- 전체 영역을 전처리 없이 두 영상의 복셀 간 상관도를 측정하는 방식임.
- 상대적으로 정확도가 높은 대신, 모든 복셀을 대상으로 유사도를 평가해야 하므로 수행 시간이 오래 걸림.
- 정합 방식:
- 중심축 및 모멘트 정합(principal axes and moments based method): 명암도 정보를 대표적인 스칼라 값이나 방향 등으로 축소하여 적용하는 방식임.
- 명암도 기반 정합(intensity-based registration): 전체 영상 정보를 사용하며, 동일 모달리티 영상 간에는
- SSD(Sum of Squares of Intensity Differences)
- 상관계수(Correlation)
를 사용하고, 서로 다른 모달리티 영상 간에는 - 조인트 엔트로피(Joint Entropy)
- 상호정보(Mutual Information)
를 사용하여 정합을 수행함.
기존 Image Registration과 학습 기반 접근 방식
- 기존의 image registration은 각 paired image에 대해 objective function을 최적화해야 하므로 대규모 데이터셋에서는 계산 비용이 큼.
- 최근 학습 기반 방법에서는 registration을 하나의 함수로 보고, 입력된 paired image를 기반으로 align된 image를 생성하는 방식으로 변형됨.
- CNN을 활용하여 registration 함수를 parameter화하고, 이미지 세트에서 신경망의 parameter를 최적화함.
VoxelMorph의 주요 학습 방법
(i) Unsupervised learning 방식으로 image 간의 matching objective function을 최적화함.
(ii) Auxiliary segmentation map을 학습에 활용하여 정합 성능을 향상시킴.
- 제안된 unsupervised model은 기존 SOTA 방법들과 비교했을 때 경쟁력 있는 결과를 제공하며, 연산 속도가 빠름.
- 추가적인 auxiliary data를 활용할 경우 registration 성능이 향상됨.
1. Introduction
Volume (3D 의료 영상의 개념)

- 의료 영상 처리 및 컴퓨터 비전에서 volume은 3D 데이터를 의미하며, 여러 개의 2D 단면(슬라이스)을 결합하여 형성됨.
- CT(컴퓨터 단층촬영) 및 MRI(자기공명영상) 데이터를 포함하며, 각 voxel(복셀, volume element)은 3D 공간 내에서 작은 단위를 의미함.
Deformable Registration (변형 정합)
- 두 개의 3D MRI 뇌 스캔과 같은 이미지 쌍 사이의 비선형적인 대응 관계를 설정하는 과정임.
- 기존 방법은 유사한 voxel을 정렬하고 registration mapping에 대한 제약을 설정하는 방식으로 최적화됨.
- 그러나 이러한 방식은 계산 비용이 크고, 속도가 느려 실제 임상 환경에서 적용하기 어려운 단점이 있음.
VoxelMorph의 접근 방식
- Learning-based registration 기법을 활용하여 volume 집합으로부터 매개변수화된 registration 함수를 학습함.
- CNN 기반 모델을 사용하여 n-D input paired volume을 입력받고, 한 volume의 모든 voxel을 다른 volume에 매핑하는 함수를 구함.
- 새로운 테스트 이미지 쌍에 대해 기존 방식처럼 복잡한 최적화를 수행하지 않고, 학습된 함수를 적용하여 빠르게 registration을 수행할 수 있도록 함.
- GPU뿐만 아니라 CPU에서도 신속한 정합이 가능하며, 다양한 loss function을 채택할 수 있는 framework임.
2. Background

기존 Volume Registration 방식
- 한 개의 이미지(소스 이미지)를 두 번째 이미지(타겟 이미지)에 맞게 변형하는 방식임.
- 변형 정합을 수행하면, 스캔 간 구조 비교가 가능하여 집단 간 차이 분석 및 질병 진행 과정 추적에 활용될 수 있음.
- 기존 방식은 Affine Transformation(전역 정렬) 후, Deformable Transformation(자유도가 높은 변형)을 수행하는 2단계 과정을 거침.
기존 Registration의 최적화 접근 방식

- 대부분의 변형 정합 알고리즘은 에너지 함수를 기반으로 최적화되며,
- 고정 이미지(f)
- 이동 이미지(m)
- 두 이미지를 정렬하는 registration field(ϕ)
를 활용하여 변환을 반복적으로 최적화함.
- 최적화 문제의 전체 손실 함수는
- 이미지 유사성 손실 함수 (L_sim)
- 변형 필드의 정규화 손실 함수 (L_smooth)
를 포함함.
3. Related Work
A. Medical Image Registration (Non-learning-based)
- 기존 연구들은 Displacement Vector Field 최적화를 기반으로 다양한 방법을 개발함.
- 주요 알고리즘:
- Elastic Type Model
- Statistical Parametric Mapping
- B-splines
- Discrete Methods
- Demons Algorithm
- 또한, 위상 보존을 위해 Diffeomorphic Transforms(다형 변환) 및 LDDMM(Large Deformation Diffeomorphic Metric Mapping) 같은 기법이 존재함.
- 하지만 이러한 접근법들은 각 image pair마다 에너지 함수를 최적화해야 하므로 속도가 느림.
- 일부 GPU 기반 알고리즘이 성능을 개선했으나, 여전히 GPU 의존성이 높음.
B. Medical Image Registration (Learning-based)
- 최근 연구들은 신경망을 활용하여 image registration을 학습하는 방식을 개발함.
- 기존 연구들은 대부분 Supervised Learning 기반으로,
- 기존 registration 방법으로 생성된 Warp Field(Ground Truth)를 학습 데이터로 사용함.
- Ground Truth Warp Field에 의존하므로, 데이터 수집이 어려울 수 있으며, 학습 가능한 변형 유형이 제한적임.
- 이에 비해 VoxelMorph는 Unsupervised Learning 방식을 채택하여,
- 필요 시 해부학적 segmentation 데이터를 추가 학습 정보로 활용 가능.
- 기존 Unsupervised Learning 기반 방법들은
- 3D 전체 volume 대신 3D 서브영역이나 2D 슬라이스만 학습 가능했으며,
- 작은 변형(small deformations)만 지원하는 한계가 있었음.
Warp Field (변형 필드)의 개념
- 이미지 정합에서 Warp Field는 이동 이미지(m)를 고정 이미지(f)에 맞춰 정렬하기 위한 픽셀(또는 복셀) 단위 변형을 의미함.
- 각 픽셀(복셀)의 좌표를 새로운 위치로 매핑하는 역할을 하며, 변형 정보 전체를 Warp Field라고 부름.
- VoxelMorph는 Warp Field를 예측하기 위해 학습된 함수 ϕ를 활용하여,
- 이동 이미지 m을 고정 이미지 f와 정렬하도록 변형함.
- 수식 표현: $$\phi(x) = x + u(x)$$ 여기서 u(x)는 각 좌표 x에 대해 적용될 Displacement(변위)를 의미함.
C. 2D Image Alignment
- Optical Flow Algorithm(광학 흐름 알고리즘)은 두 개의 2D 이미지 간의 작은 Displacement를 나타내는 Dense Displacement Vector Field를 반환함.
- 기존에는 Variational Method(변분법)을 사용하여 최적화 문제를 해결했으며,
- Feature-based Matching(기능 기반 매칭) 기법을 활용하여 대규모 Displacement를 처리함.
- 최근에는 신경망을 활용한 2D Image Registration 기법이 등장함.
- 일부 연구들은 Supervised Learning 방식을 활용하지만,
- Unsupervised Learning 기반 방법도 개발됨.
- VoxelMorph는 이러한 2D Registration 기법을 확장하여 n-D Volume Registration에 적용,
- Segmentation 데이터를 학습에 활용하여 정확도를 향상시키는 방법을 제공함.
4. Method

A. VoxelMorph CNN Architecture

- U-Net 기반 CNN 모델을 사용하여 registration을 수행함.
- 입력 데이터: Volume Pair (f, m)
- 고정 이미지 f
- 이동 이미지 m
- 두 이미지를 결합하여 2채널 3D 이미지로 입력
- 구조:
- Encoder:
- 3D Stride Convolution을 사용하여 입력 차원을 절반으로 줄임.
- Decoder:
- Upsampling을 사용하여 원래 크기로 복원함.
- Skip Connection:
- Encoder의 특징을 Decoder로 전달하여 registration 성능을 향상시킴.
- Encoder:
B. Spatial Transformation Function

- Warp Field(ϕ)를 학습하기 위해 공간 변환 함수를 사용함.
- 각 Voxel p에 대해 새로운 위치 p'을 계산하며,
- 8개의 인접 Voxel을 선형 보간(Linear Interpolation)하여 최종 변형된 이동 이미지 m ∘ ϕ를 구함.
- 이 과정은 미분 가능(Differentiable)하도록 설계되어 있으며,
- 신경망이 역전파(Backpropagation)를 통해 손실 함수를 최적화할 수 있도록 구성됨.
C. Loss Functions
VoxelMorph는 Unsupervised Loss Function과 Auxiliary Data Loss Function을 적용하여 registration 성능을 향상시킴.
(1) Unsupervised Loss Function

- $L_{sim}$ (이미지 유사성 손실):
- 고정 이미지 f와 변형된 이동 이미지 m ∘ ϕ 간의 유사성을 측정함.
- Mean Squared Error (MSE)
- Local Cross-Correlation (CC)
- $L_{smooth}$ (변형 필드의 매끄러움 손실):

- 변형이 너무 급격하게 변하지 않도록 필드의 공간적 변화율을 최소화함.
- u = ϕ - Id를 변형 필드의 변위(Displacement)로 정의함.
- ∇u(p)는 변형 필드의 공간적 그래디언트를 나타냄.
(2) Auxiliary Data Loss Function
- 추가적인 해부학적 segmentation 정보를 활용하여 registration 성능을 향상시킴.

- $L_{seg}$ (Segmentation Alignment Loss):
- Dice Score를 기반으로 각 해부학적 구조가 얼마나 정확히 정합되었는지를 평가함.
- Dice Score가 1에 가까울수록 더 정확한 정합을 의미함.
$$L_{seg} (s_f, s_m ∘ ϕ) = -\frac{1}{K} \sum_{k=1}^{K} Dice(s_f^k, (s_m ∘ ϕ)^k)$$
- s_f: 고정 이미지의 Segmentation Map
- s_m: 이동 이미지의 Segmentation Map
5. Experiments
A. Experimental Setup

- Dataset:
- 총 3,731개 T1 가중 뇌 MRI 스캔을 사용하여 실험 진행함.
- Baseline 비교 모델:
- 기존 ANTs, NiftyReg와 비교하여 성능 평가함.
- 평가 지표:
- Dice Score (정합된 해부학적 구조 간의 겹침 정도)
B. Atlas-based Registration

- 특정 Atlas Image를 기준으로 다양한 대상 이미지를 정렬하는 방식임.
- VoxelMorph는 기존 방법 대비 CPU 및 GPU에서 빠르게 동작하면서도 유사한 성능을 제공함.
C. Regularization Analysis

- 정규화 매개변수 λ 변화에 따른 성능 분석을 진행함.
- Dice Score가 λ 값 변화에 따라 크게 변하지 않음을 확인하여,
- VoxelMorph가 λ 선택에 대해 비교적 Robust함을 증명함.
D. Training Set Size 영향 분석

- 데이터셋 크기가 클수록 성능이 향상됨.
E. Subject-to-Subject Registration

- Atlas 없이 임의의 두 뇌 MRI Volume 간 Registration을 테스트함.
- 기존 방법 대비 VoxelMorph가 훨씬 빠르게 Registration을 수행함.
'Paper Review > Medical AI' 카테고리의 다른 글
| [OUTTA Alpha팀 논문 리뷰 요약] Part 10-7. MAISI: Medical AI for Synthetic Imaging(3D CT 이미지 생성 모델) (0) | 2025.02.09 |
|---|

